Tirosina hidroxilasa
| Tirosina hidroxilasa | ||||||
|---|---|---|---|---|---|---|
 | ||||||
| Estructuras disponibles | ||||||
| PDB | Buscar ortólogos: PDBe, RCSB Lista de códigos PDB 2XSN , 4J6S Estructuras enzimáticas RCSB PDB, PDBe, PDBsum | |||||
| Identificadores | ||||||
| Símbolos | TH (HGNC: 11782) DYT14; DYT5b; TYH | |||||
| Identificadores externos | Bases de datos de enzimas IntEnz: entrada en IntEnz BRENDA: entrada en BRENDA ExPASy: NiceZime view KEGG: entrada en KEEG PRIAM: perfil PRIAM ExplorEnz: entrada en ExplorEnz MetaCyc: vía metabólica | |||||
| Número EC | 1.14.16.2 | |||||
| Locus | Cr. 11 p15.5 | |||||
| ||||||
| Patrón de expresión de ARNm | ||||||
 | ||||||
| Más información | ||||||
| Ortólogos | ||||||
| Especies |
| |||||
| Entrez |
| |||||
| Ensembl |
| |||||
| UniProt |
| |||||
| RefSeq (ARNm) |
| |||||
| RefSeq (proteína) NCBI |
| |||||
| Ubicación (UCSC) |
| |||||
| PubMed (Búsqueda) |
| |||||
| ||||||
[editar datos en Wikidata] | ||||||
La tirosina hidroxilasa o tirosina 3-monooxigenasa (EC 1.14.16.2) es la enzima responsable de catalizar la conversión del aminoácido L-tirosina a dihidroxifenilalanina (DOPA). La DOPA es el precursor de la dopamina, que a su vez es también el precursor de la noradrenalina y la adrenalina. En humanos, la tirosina hidroxilasa es codificada por el gen TH.[1]
Reacción catalizada
La enzima, una oxigenasa, se encuentra en el citosol de todas las células que sintetizan catecolaminas. La reacción inicial es el paso limitante en la producción de catecolaminas.
La enzima es altamente específica, no aceptando derivados indólicos (lo cual es frecuente en muchas otras enzimas involucradas en la producción de catecolaminas).
 +
+ 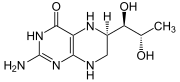 + O2 →
+ O2 →  +
+ 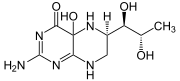 + H2O
+ H2O

Estructura y características de la enzima
La tirosina hidroxilasa (TH), también denominada tirosina 3-monooxigenasa, pertenece a la familia de hidroxilasas de aminoácidos aromáticos. Estas enzimas utilizan tetrahidrobiopterina como coenzima y hierro (II) no unido a un grupo hemo como cofactor.[2] La TH tiene una masa atómica de 240 kDa y está formada por cuatro unidades idénticas de 60kDa (homotetrámero). El gen de la TH en humanos está formado por 14 exones separados por 13 intrones, que tras su traducción da lugar a 4 isoformas distintas que difieren en su estructura peptídica.[3]
En cada subunidad del tetrámero se pueden identificar dos dominios, uno catalítico y uno regulador. En dominio catalítico que corresponde al extremo C-terminal es el más conservado en la evolución y donde se encuentra unido el cofactor, y donde interaccionan la coenzima y el sustrato durante la catálisis. El dominio regulador está en el extremo N-terminal y tienen un efecto inhibidor sobre la actividad enzimática. Esta inhibición desaparece cuando se activa la enzima mediante fosforilación de residuos de serina situados en Ser-8, Ser-19, Ser-31, Ser-40 y Ser-153. Estas fosforilaciones se llevan a cabo por proteína quinasas que siguen distintos mecanismos de regulación.[3]
Importancia clínica
La tirosina hidroxilasa se puede inhibir con la α-metiltirosina (Metirosina), aunque su administración no sea un medio eficaz para regular la síntesis de noradrenalina. Este fármaco se usa poco, pero se ha utilizado con éxito en el tratamiento del feocromocitoma y de la hipertensión.
La tirosina hidroxilasa es un autoantigeno en una enfermedad autoinmune conocida como Síndrome poliglandular autoinmune (tipo I).[4]
Como ejemplos de otros inhibidores más antiguos mencionados en la litura tenemos oudenone[5] y aquayamicina.[6]
Referencias
- ↑ Nagatsu T (1995). «Tyrosine hydroxylase: human isoforms, structure and regulation in physiology and pathology». Essays Biochem. 30: 15-35. PMID 8822146.
- ↑ Haavik, J., Toska, K., Tyrosine Hydroxylase and Parkinson’s Disease. Molecular Neurobiology, 1998. 16(3) :p.285-309.
- ↑ a b Goldstein, M., Lieberman, A., The role of the regulatory enzymes of catecholamine synthesis in Parkinson’s disease. NEUROLOGY, 1992. 42(4) :p.8-10.
- ↑ Hedstrand H, Ekwall O, Haavik J, Landgren E, Betterle C, Perheentupa J, Gustafsson J, Husebye E, Rorsman F, Kämpe O (enero de 2000). «Identification of tyrosine hydroxylase as an autoantigen in autoimmune polyendocrine syndrome type I». Biochem. Biophys. Res. Commun. 267 (1): 456-61. PMID 10623641. doi:10.1006/bbrc.1999.1945.
- ↑ Ono M, Okamoto M, Kawabe N, Umezawa H, Takeuchi T (marzo de 1971). «Oudenone, a novel tyrosine hydroxylase inhibitor from microbial origin». J. Am. Chem. Soc. 93 (5): 1285-6. PMID 5545929. doi:10.1021/ja00734a054.
- ↑ Ayukawa S, Takeuchi T, Sezaki M, Hara T, Umezawa H (mayo de 1968). «Inhibition of tyrosine hydroxylase by aquayamycin». J. Antibiot. 21 (5): 350-3. PMID 5726288.
- Pharmacology 5ª Ed, por Rang, Dale Ritter y Moore
Léase también
- Masserano JM, Weiner N (1983). «Tyrosine hydroxylase regulation in the central nervous system.». Mol. Cell. Biochem. 53-54 (1-2): 129-52. PMID 6137760. doi:10.1007/BF00225250.
- Meloni R, Biguet NF, Mallet J (2002). «Post-genomic era and gene discovery for psychiatric diseases: there is a new art of the trade? The example of the HUMTH01 microsatellite in the Tyrosine Hydroxylase gene.». Mol. Neurobiol. 26 (2-3): 389-403. PMID 12428766. doi:10.1385/MN:26:2-3:389.
- Joh TH, Park DH, Reis DJ (1979). «Direct phosphorylation of brain tyrosine hydroxylase by cyclic AMP-dependent protein kinase: mechanism of enzyme activation.». Proc. Natl. Acad. Sci. U.S.A. 75 (10): 4744-8. PMC 336196. PMID 33381. doi:10.1073/pnas.75.10.4744.
- Haycock JW, Ahn NG, Cobb MH, Krebs EG (1992). «ERK1 and ERK2, two microtubule-associated protein 2 kinases, mediate the phosphorylation of tyrosine hydroxylase at serine-31 in situ.». Proc. Natl. Acad. Sci. U.S.A. 89 (6): 2365-9. PMC 48658. PMID 1347949. doi:10.1073/pnas.89.6.2365.
- Haycock JW (1990). «Phosphorylation of tyrosine hydroxylase in situ at serine 8, 19, 31, and 40.». J. Biol. Chem. 265 (20): 11682-91. PMID 1973163.
- Craig SP, Buckle VJ, Lamouroux A, et al. (1986). «Localization of the human tyrosine hydroxylase gene to 11p15: gene duplication and evolution of metabolic pathways.». Cytogenet. Cell Genet. 42 (1-2): 29-32. PMID 2872999. doi:10.1159/000132246.
- Grima B, Lamouroux A, Boni C, et al. (1987). «A single human gene encoding multiple tyrosine hydroxylases with different predicted functional characteristics.». Nature 326 (6114): 707-11. PMID 2882428. doi:10.1038/326707a0.
- Kaneda N, Kobayashi K, Ichinose H, et al. (1987). «Isolation of a novel cDNA clone for human tyrosine hydroxylase: alternative RNA splicing produces four kinds of mRNA from a single gene.». Biochem. Biophys. Res. Commun. 146 (3): 971-5. PMID 2887169. doi:10.1016/0006-291X(87)90742-X.
- Kobayashi K, Kaneda N, Ichinose H, et al. (1987). «Isolation of a full-length cDNA clone encoding human tyrosine hydroxylase type 3.». Nucleic Acids Res. 15 (16): 6733. PMC 306135. PMID 2888085. doi:10.1093/nar/15.16.6733.
- O'Malley KL, Anhalt MJ, Martin BM, et al. (1988). «Isolation and characterization of the human tyrosine hydroxylase gene: identification of 5' alternative splice sites responsible for multiple mRNAs.». Biochemistry 26 (22): 6910-4. PMID 2892528. doi:10.1021/bi00396a007.
- Le Bourdellès B, Boularand S, Boni C, et al. (1988). «Analysis of the 5' region of the human tyrosine hydroxylase gene: combinatorial patterns of exon splicing generate multiple regulated tyrosine hydroxylase isoforms.». J. Neurochem. 50 (3): 988-91. PMID 2892893. doi:10.1111/j.1471-4159.1988.tb03009.x.
- Ginns EI, Rehavi M, Martin BM, et al. (1988). «Expression of human tyrosine hydroxylase cDNA in invertebrate cells using a baculovirus vector.». J. Biol. Chem. 263 (15): 7406-10. PMID 2896667.
- Kobayashi K, Kaneda N, Ichinose H, et al. (1988). «Structure of the human tyrosine hydroxylase gene: alternative splicing from a single gene accounts for generation of four mRNA types.». J. Biochem. 103 (6): 907-12. PMID 2902075.
- Coker GT, Vinnedge L, O'Malley KL (1989). «Characterization of rat and human tyrosine hydroxylase genes: functional expression of both promoters in neuronal and non-neuronal cell types.». Biochem. Biophys. Res. Commun. 157 (3): 1341-7. PMID 2905129. doi:10.1016/S0006-291X(88)81022-2.
- Vulliet PR, Woodgett JR, Cohen P (1984). «Phosphorylation of tyrosine hydroxylase by calmodulin-dependent multiprotein kinase.». J. Biol. Chem. 259 (22): 13680-3. PMID 6150037.
- Zhou QY, Quaife CJ, Palmiter RD (1995). «Targeted disruption of the tyrosine hydroxylase gene reveals that catecholamines are required for mouse fetal development.». Nature 374 (6523): 640-3. PMID 7715703. doi:10.1038/374640a0.
- Lüdecke B, Bartholomé K (1995). «Frequent sequence variant in the human tyrosine hydroxylase gene.». Hum. Genet. 95 (6): 716. PMID 7789962. doi:10.1007/BF00209496.
- Lüdecke B, Dworniczak B, Bartholomé K (1995). «A point mutation in the tyrosine hydroxylase gene associated with Segawa's syndrome.». Hum. Genet. 95 (1): 123-5. PMID 7814018. doi:10.1007/BF00225091.
- Knappskog PM, Flatmark T, Mallet J, et al. (1996). «Recessively inherited L-DOPA-responsive dystonia caused by a point mutation (Q381K) in the tyrosine hydroxylase gene.». Hum. Mol. Genet. 4 (7): 1209-12. PMID 8528210. doi:10.1093/hmg/4.7.1209.
 Datos: Q420766
Datos: Q420766











